OpenMM 是一个用于分子模拟的高性能工具包。可以将其用作库调用或作为独立程序运行。
此工具包还包含适用于 Python、C、C++ 与 Fortran 的语言绑定。
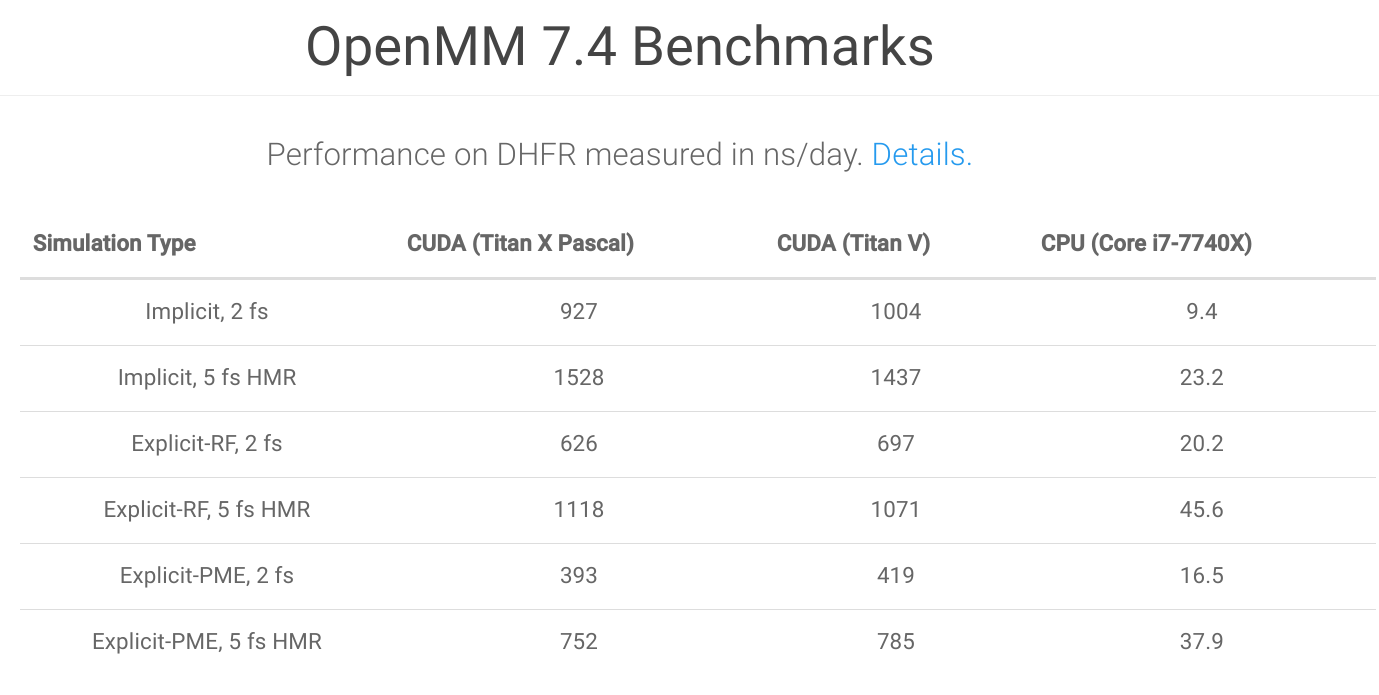
这一工具包具有极高的灵活性与速度,通过 GPU 加速以及 AMD、NVIDIA 和 Intel 集成 GPU 的优化实现了卓越的性能。
基准测试:
快速使用(Python):
from simtk.openmm.app import *
from simtk.openmm import *
from simtk.unit import *
from sys import stdout
pdb = PDBFile(\'input.pdb\')
forcefield = ForceField(\'amber99sb.xml\', \'tip3p.xml\')
system = forcefield.createSystem(pdb.topology, nonbondedMethod=PME, nonbondedCutoff=1*nanometer, constraints=HBonds)
integrator = LangevinIntegrator(300*kelvin, 1/picosecond, 0.002*picoseconds)
simulation = Simulation(pdb.topology, system, integrator)
simulation.context.setPositions(pdb.positions)
simulation.minimizeEnergy()
simulation.reporters.append(PDBReporter(\'output.pdb\', 1000))
simulation.reporters.append(StateDataReporter(stdout, 1000, step=True, potentialEnergy=True, temperature=True))
simulation.step(10000)
© 版权声明
文章版权归作者所有,未经允许请勿转载。
THE END


















暂无评论内容